No post Por que evitar for loops em R? mostramos que for loops são ineficientes em R. Para isso, tal abordagem foi comparada a outras alternativas (funções do purrr e da família apply), dentre elas opções que usam paralelização. No entanto, no exemplo utilizado, havia um número grande de iterações muito simples, o que não gerou ganho de eficiência nos casos paralelizados.
Neste post, vamos apresentar um caso em que a paralelização apresenta ganhos de eficiência significativos. Para isso, vamos treinar um modelo Random Forest (Floresta Aleatória) de classificação para prever as espécies de flores baseando-se em quatro características: largura e comprimento das pétalas e sépalas.
Os dados
Primeiramente, vamos carregar os dados. Não é necessário o uso de nenhum pacote para isso. Basta utilizar o código abaixo.
data(iris)Explorando os dados, vemos que existem 150 observações na base de dados. Também, notamos que cada uma das espécies aparece a mesma quantidade de vezes, 50 vezes cada. A largura e o comprimento das pétalas e sépalas estão em centímetro. O código abaixo apresenta o resumo dos dados.
summary(iris)
Sepal.Length Sepal.Width Petal.Length Petal.Width
Min. :4.300 Min. :2.000 Min. :1.000 Min. :0.100
1st Qu.:5.100 1st Qu.:2.800 1st Qu.:1.600 1st Qu.:0.300
Median :5.800 Median :3.000 Median :4.350 Median :1.300
Mean :5.843 Mean :3.057 Mean :3.758 Mean :1.199
3rd Qu.:6.400 3rd Qu.:3.300 3rd Qu.:5.100 3rd Qu.:1.800
Max. :7.900 Max. :4.400 Max. :6.900 Max. :2.500
Species
setosa :50
versicolor:50
virginica :50
Fundamentos introdutórios do Random Forest
Como dissemos anteriormente, treinar um modelo de classificação é apenas o meio utilizado para mostrar a utilidade da paralelização. No entanto, já que vamos utilizar o Random Forest para isso, vale a pena uma breve introdução.
O Random Forest é um tipo de modelo usado tanto para classificação quanto para regressão, que é baseado no que chamamos de árvores de decisão (em inglês, decision trees). Árvores de decisão nada mais são do que um processo sequencial de seleções binárias (ou seja, verdadeiro ou falso, sim ou não…) no qual cada nó da árvore representa uma “pergunta” relacionada a uma das variáveis explicativas. Ao seguir todo o caminho da árvore, atingimos uma de suas folhas a qual indica a classificação prevista pelo modelo. A ordem das variáveis para a realização das “perguntas” considera o quão informativa essa variável é, ou seja, o quanto os valores dessa variável divide a base de dados em conjuntos homogêneos.
Confuso? Vamos entender melhor com exemplos. Vocês se lembram de um jogo de criança chamado “Cara a Cara”? Deem uma olhada abaixo que provavelmente se lembrarão.

O jogo consistem em tentar adivinhar qual personagem o seu adversário tirou da pilha de cartas. Para isso, fazemos perguntas relacionadas às características dos personagens e, caso a resposta seja “sim”, eliminamos todos os personagens que não possuem aquele característica (e vice-versa). Suponha, então, que eu tenha tirado o personagem João. Meu adversário pergunta se o meu personagem usa chapéu. Como eu irei responder “não”, ele eliminará todos os personagens que usam chapéu (o Beto, a Clara, o Henrique e a Maria). Esse processo será repetido até um dos jogadores conseguir identificar a carta do oponente.
Vamos sair da analogia e ir para o exemplo dos dados das flores que já carregamos em nosso computador. Após treinarmos o modelo, uma árvore foi construída com “perguntas” específicas em seus nós relacionadas às características das flores. Uma das perguntas pode ser: o comprimento da pétala é maior do que 3,3 cm? Ou então: a largura da sépala é menor do que 2,8 cm? Vejamos a imagem abaixo para termos uma ideia mais clara (os valores são apenas ilustrativos).
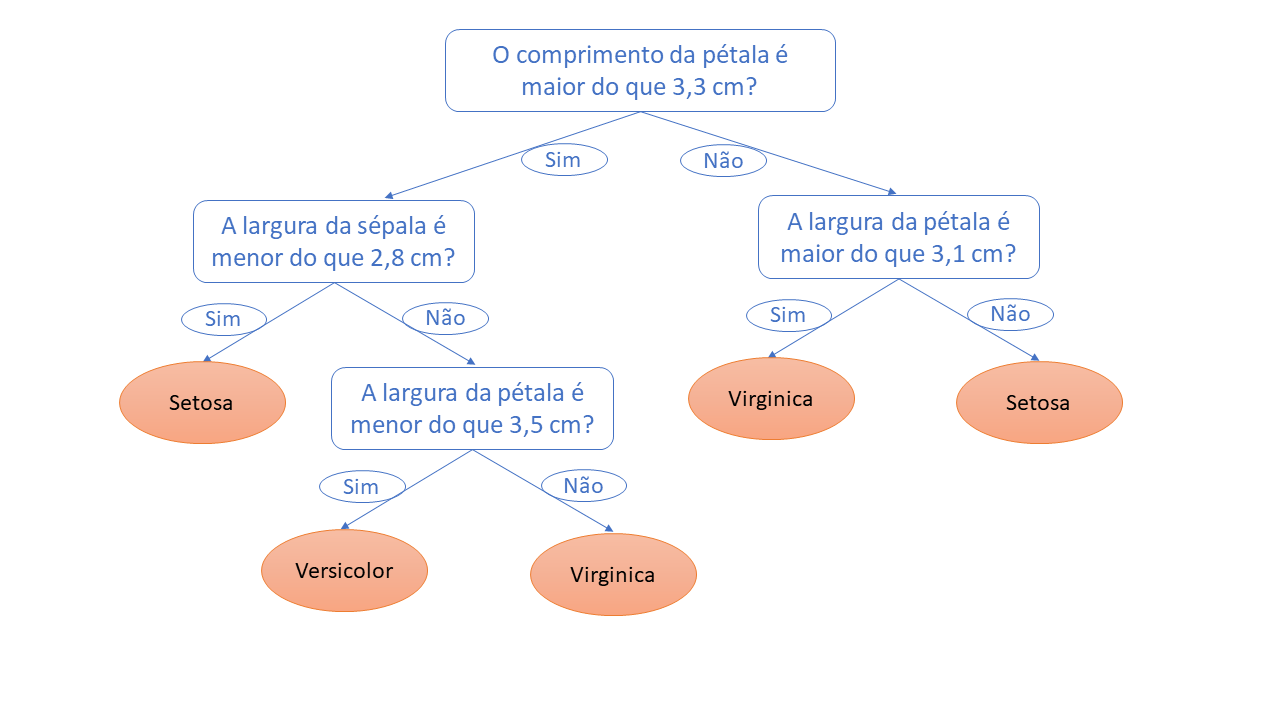
Agora que entendemos como uma árvore de decisão funciona, fica mais fácil patirmos para o Random Forest. Random Forest nada mais é um conjunto de árvores de decisão. Mais especificamente, este modelo combina várias árvores de decisão, treinando cada uma delas com um conjunto diferente de observações definido de forma aleatória. Além disso, cada árvore utiliza um número limitado de características (variáveis) também escolhidas aleatoriamente. No fim, as previsões são feitas considerando uma média das previsões de cada árvore individualmente.
O Random forest utilizando o pacote caret
O pacote caret funciona como uma interface para diversos outros pacotes muito utilizados em trabalhos de aprendizado de máquina (machine learning) e estatística. Duas de suas principais funções são: train() e trainControl(). A função train(), como o nome diz, é responsável por treinar o modelo. Nela, indicamos os dados que vamos usar, a variável dependente e as variáveis independentes, o método a ser utilizado (no nosso caso o método é “rf” de Random Forest), e os parâmentros do método que iremos testar.
A função trainControl(), por sua vez, indica se usaremos validação cruzada (cross validation), quantos subconjuntos (folds) serão utilizados, quantos repetições da validação cruzada serão realizadas (se for o caso), entre outros aspectos.
Nest post, para avaliar a eficiência da paralelização, vamos testar diferentes combinações de parâmentros do modelo para descobrir qual especificação gera melhores resultados, o que em ciência de dados é chamado de grid search. Os parâmetros para os quais diferentes valores são testados são o número de variáveis independentes usadas em cada uma das árvores, a quantidade total de árvores no modelo e o número mínimo de observações classificadas em cada uma das folhas das árvores durante o treinamento. É importante frisar que o caret nos permite realizar o grid search e a paralelização de forma direta, como argumentos de suas funções. No entanto, para fins didáticos, faremos o processo “manualmente”.
Quebrando os dados em amostra para treinamento e para teste
Após treinarmos um modelo, precisamos testá-lo para saber o quão boas são suas previsões. Se usarmos o mesmo conjunto de dados para treinar e para testar estaremos trapaceando. O teste precisa ser feito em um conjunto de dados que não foi visto previamente pelo modelo. Como fazemos isso? Simples, separamos parte dos dados para servirem como teste e utilizamos o restante para treinar o modelo. Como pode ser visto no código abaixo, usaremos 90% dos dados para treinamento e 10% para teste. Outro detalhe importante é o uso de uma amostra estratificada, ou seja, os dados utilizados para teste possuem a mesma (ou bem similar) proporção de observações de cada classe (espécie).
# Carregar dplyr para manipulação dos dados
library(dplyr)
# Definir seed para reprodutibilidade
set.seed(123)
# Definir dados de treinamento e teste usando amostra estratificada
iris <- iris %>%
mutate(indice_linha = row_number())
treinamento <- iris %>%
group_by(Species) %>%
sample_frac(., size = 0.9)
teste <- iris %>%
filter(!indice_linha %in% treinamento$indice_linha)
treinamento <- treinamento %>% select(-indice_linha)
teste <- teste %>% select(-indice_linha)Definindo a função para selecionar a “melhor” combinação de parâmetros
Agora que temos nossa base para treinamento, vamos criar uma função para treinar o nosso Random Forest e encontrar a combinação de parâmentros que faz com que a acurácia das previsões seja a maior possível. Primeiramente, iremos criar uma lista de seeds que são usadas em cada uma das iterações da validação cruzada de forma a possibilitar que os resultados de todos que rodem o mesmo código sejam iguais mesmo considerando a aleatoriedade que existe em diferentes momentos do processo de treinamento.
# Carregar caret
library(caret)
# Definir seeds
seeds <- lapply(c(1:30), function(i){sample.int(1000, 27)})
seeds[[31]] <- sample.int(1000, 1)Como queremos investigar qual é a combinação de parâmetros que leva a melhores resultados, precisamos definir todas as combinações que queremos verificar. Fazemos isso usando a função expand.grid() e incluindo todos os valores de cada parâmetro que nos interessa. No nosso caso, testaremos os valores 1, 2 e 3 para o número de variáveis explicativas usadas em cada uma das árvores; 300, 400 e 500 para a quantidade total de árvores no modelo; e 5, 10 e 15 para o número mínimo de observações classificadas em cada uma das folhas das árvores durante o treinamento.
grid_completo <- expand.grid(mtry = c(1, 2, 3),
ntree = c(300, 400, 500),
nodesize = c(5, 10, 15))Podemos, então, definir a função trainControl(). Nela, indicamos que vamos realizar uma validação cruzada com repetições (repeatedcv), na qual serão considerados 10 subconjuntos em cada uma das três validações cruzadas. O uso do repeatedcv é interessante quando não há muitos dados para treinamento.
fit_control <- trainControl(method = "repeatedcv",
number = 10,
repeats = 3,
seeds = seeds)Finalmente, definimos a função que efetivamente treina o modelo testando as combinações de parâmentros definidas. Essa função recebe como o argumento cada um dos parâmetros do grid e gera um data frame com a acurácia para cada uma das combinações.
rf_cv <- function(mtry_val, ntree_val, nodesize_val) {
mtry_temp <- data.frame(.mtry = mtry_val)
set.seed(123)
rf_fit <- train(Species ~ .,
data = treinamento,
method = "rf",
trControl = fit_control,
tuneGrid = mtry_temp,
ntree = ntree_val,
nodesize = nodesize_val)
resultados <- rf_fit$results %>%
mutate(ntree = ntree_val,
nodesize = nodesize_val) %>%
select(mtry, ntree, nodesize, Accuracy)
return(resultados)
}Finalmente a paralelização
Vamos voltar para o objetivo principal deste post. Queremos verificar se a paralelização realmente traz ganhos de desempenho. Como o grid que construímos possui 27 diferentes combinações de parâmentros, precisamos testar cada uma delas no modelo definido acima. Podemos fazer isso de forma sequencial ou paralelizada. Sequencialmente, uma combinação só começa a ser testada após o término da anterior. Usando a abordagem paralelizada, vários núcleos do computador trabalham simultaneamente, ou seja, cada um deles fica responsável por um conjunto de combinações. Vamos aos testes!
Como mostrado no post Por que evitar for loops em R?, é sempre bom evitarmos for loops em R. Seguindo essa boa prática, aqui usaremos o pacote purrr e a família apply e suas versões paralelizadas (pacote furrr e future.apply). Primeiramente, vamos carregar os pacotes necessários.
library(future.apply)
library(purrr)
library(furrr)
library(caret)
plan(multiprocess)Vale uma atenção especial para a função plan(). Ela é uma função do pacote future que é responsável por planejar como as tarefas serão executadas. Quando queremos paralelizar, duas estratégias são possíveis. Uma é a chamada multicore, que usa mais de um núcleo do computador para realizar a tarefa. A outra estratégia é utilizar mais de uma sessão de R para realizar a tarefa de forma paralelizada, que é chamada de multisession. Nem todos os sistemas operacionais suportam o multicore, como o Windows por exemplo. Para não precisarmos saber todos os casos, uma alternativa é usar a estratégia multiprocess, já que ela utiliza o multicore, se suportado; caso contrário, ela usa o multisession.
Agora, podemos rodar nosso modelo Random Forest usando as quatro formas que serão testadas.
# PURRR
purrr_tempo <- system.time({
purrr_df <- pmap_df(grid_completo, rf_cv)
})
# FURRR
furrr_tempo <- system.time({
furrr_df <- future_pmap_dfr(grid_completo, rf_cv)
})
# APPLY
apply_tempo <- system.time({
apply_df <- mapply(
rf_cv,
mtry_val = grid_completo$mtry,
ntree_val = grid_completo$ntree,
nodesize_val = grid_completo$nodesize
)
mtry_val <- unlist(apply_df)[c(seq(from = 1, to = 108, by = 4))]
ntree_val <- unlist(apply_df)[c(seq(from = 2, to = 108, by = 4))]
nodesize_val <- unlist(apply_df)[c(seq(from = 3, to = 108, by = 4))]
accuracies <- unlist(apply_df)[c(seq(from = 4, to = 108, by = 4))]
apply_df <- data.frame(mtry = mtry_val, ntree = ntree_val,
nodesize = nodesize_val, Accuracy = accuracies)
})
# FUTURE APPLY
future_apply_tempo <- system.time({
future_apply_df <- future_mapply(
rf_cv,
mtry_val = grid_completo$mtry,
ntree_val = grid_completo$ntree,
nodesize_val = grid_completo$nodesize
)
mtry_val <- unlist(future_apply_df)[c(seq(from = 1, to = 108, by = 4))]
ntree_val <- unlist(future_apply_df)[c(seq(from = 2, to = 108, by = 4))]
nodesize_val <- unlist(future_apply_df)[c(seq(from = 3, to = 108, by = 4))]
accuracies <- unlist(future_apply_df)[c(seq(from = 4, to = 108, by = 4))]
future_apply_df <- data.frame(mtry = mtry_val, ntree = ntree_val,
nodesize = nodesize_val, Accuracy = accuracies)
})Antes de verificarmos o tempo levado por cada uma das diferentes abordagens, vamos checar se os data frames de resultados gerados são os mesmos e visualizar as primeiras linhas de um deles.
# Checar se data frames são iguais
all_equal(purrr_df, furrr_df)
[1] TRUE
all_equal(furrr_df, apply_df)
[1] TRUE
all_equal(apply_df, future_apply_df)
[1] TRUE
# Ver primeiras linhas de um data frame de resultados
future_apply_df %>% arrange(-Accuracy) %>% head()
mtry ntree nodesize Accuracy
1 2 300 15 0.9478755
2 2 400 15 0.9478755
3 2 500 15 0.9478755
4 2 300 5 0.9476923
5 3 300 5 0.9476923
6 3 400 5 0.9476923Vemos que as combinações que geraram previsões mais precisas são as que usam duas variáveis explicativas escolhidas aleatoriamente e no mínimo 15 observações classificadas em cada uma das folhas das árvores durante o treinamento. Além disso, vimos também que todas as abordagens geraram os mesmos resultados.
E, então, qual dos métodos utilizados foram mais eficientes em relação ao tempo gasto?
purrr_tempo
user system elapsed
23.46 1.10 25.13
furrr_tempo
user system elapsed
0.08 0.00 23.63
apply_tempo
user system elapsed
23.95 0.81 25.78
future_apply_tempo
user system elapsed
0.04 0.00 10.42 Realmente neste caso a paralelização foi significativamente mais rápida. Para os pacotes purrr e furrr, houve uma redução de tempo de 6%. Já para a família apply o ganho de desempenho foi ainda maior: redução de 59.6%.
Mas afinal, o quão preciso é nosso modelo?
Como dissemos anteriormente, precisamos verificar o quão precisas são as previsões de nosso modelo aplicando-o a dados não vistos anteriormente. Vamos, então, usar nosso modelo e prever as observações contidas no conjunto de dados separado para teste.
Primeiramente, vamos treinar novamente nosso modelo usando os valores dos parâmetros que geraram os melhores resultados.
# Selecionar combinação com melhores resultados
parametros_max <- future_apply_df %>% arrange(-Accuracy) %>% slice(1)
# Utilizar parâmetros para treinar Random Forest
set.seed(123)
rf_fit_final <- train(x = treinamento[, -5],
y = treinamento[, 5] %>% pull(),
method = "rf",
trControl = trainControl("none"),
tuneGrid = data.frame(.mtry = parametros_max[[1]]),
ntree = parametros_max[[2]],
nodesize = parametros_max[[3]])Agora vamos utilizar o modelo treinado para prever as espécies das flores dos dados separados para teste. Para isso, usamos a função predict(), que a partir das variáveis explicativas prevê para cada observação uma das três possíveis espécies. Em seguida usamos a função confusionMatrix() para verifcar as métricas de avaliação.
previsoes <- predict(rf_fit_final, teste[, -5])
confusionMatrix(previsoes, teste[, 5])
Confusion Matrix and Statistics
Reference
Prediction setosa versicolor virginica
setosa 5 0 0
versicolor 0 5 0
virginica 0 0 5
Overall Statistics
Accuracy : 1
95% CI : (0.782, 1)
No Information Rate : 0.3333
P-Value [Acc > NIR] : 6.969e-08
Kappa : 1
Mcnemar's Test P-Value : NA
Statistics by Class:
Class: setosa Class: versicolor Class: virginica
Sensitivity 1.0000 1.0000 1.0000
Specificity 1.0000 1.0000 1.0000
Pos Pred Value 1.0000 1.0000 1.0000
Neg Pred Value 1.0000 1.0000 1.0000
Prevalence 0.3333 0.3333 0.3333
Detection Rate 0.3333 0.3333 0.3333
Detection Prevalence 0.3333 0.3333 0.3333
Balanced Accuracy 1.0000 1.0000 1.0000Excelente! Nosso modelo foi capaz de prever todas as flores do conjunto de teste corretamente.
Conclusão
Neste post, fomos capazes de mostrar um aumento de desempenho ao utilizarmos abordagens paralelizadas para classificar espécies de flores a partir de um modelo Random Forest. A paralelização se torna ainda mais importante quando precisamos realizar uma tarefa rotineira que nos demanda muito tempo ou quando criamos algum tipo de interface que precisa gerar respostas rápidas. Aproveitamos o nosso teste sobre paralelização para entender e testar o modelo de classificação Random Forest utilizando o pacote caret. Nosso modelo foi capaz de realizar previsões excelentes.