Introdução
Escuto com frequência que for loops deveriam ser evitados em R. O motivo: eles não são tão eficientes em R como são em outras linguagens de programação. Existem, porém, formas de substituir for loops usando, por exemplo, funções da família apply, o pacote purrr e abordagens de paralelização.
Neste post vamos comparar a performance do for loop e seus substitutos por meio de um simples exercício.
Tarefa
Primeiramente, vamos carregar o pacote dplyr para montar a estrutura da tarefa e definir uma seed para fins de replicação.
library(dplyr)
set.seed(123)Agora vamos criar um data frame no qual cada uma das 3 colunas é formada por valores criados a partir de uma curva normal com média zero e desvio padrão 1.
n_linhas <- 500000
dados_base <- data.frame(A = rnorm(n_linhas), B = rnorm(n_linhas), C = rnorm(n_linhas))
head(dados_base)
A B C
1 -0.56047565 0.2620001 -1.0080707
2 -0.23017749 -1.5209594 1.3549394
3 1.55870831 0.2366280 -0.4689749
4 0.07050839 0.6603038 1.4681936
5 0.12928774 -0.2140063 0.4425564
6 1.71506499 0.1928270 0.1462031O nosso objetivo é criar uma quarta coluna onde cada elemento seja a soma dos elementos das demais colunas na mesma linha, ou seja, o elemento na primeira linha da quarta coluna é a soma dos elementos da primeira linha das colunas A, B e C.
Para entendermos melhor o uso deste exemplo, vamos tentar realizar essa tarefa criando uma nova coluna com o dplyr usando a função sum():
dados_base %>% mutate(D = sum(A, B, C)) %>% head()
A B C D
1 -0.56047565 0.2620001 -1.0080707 -1941.632
2 -0.23017749 -1.5209594 1.3549394 -1941.632
3 1.55870831 0.2366280 -0.4689749 -1941.632
4 0.07050839 0.6603038 1.4681936 -1941.632
5 0.12928774 -0.2140063 0.4425564 -1941.632
6 1.71506499 0.1928270 0.1462031 -1941.632Usando a função sum(), não conseguimos obter o resultado que desejamos. Neste caso, cada elemento da coluna D é a soma de todos os elementos de cada uma das colunas.
Uma forma de resolver isto e chegar no resultado que queremos seria:
dados_base %>% mutate(D = A + B + C) %>% head()
A B C D
1 -0.56047565 0.2620001 -1.0080707 -1.3065463
2 -0.23017749 -1.5209594 1.3549394 -0.3961975
3 1.55870831 0.2366280 -0.4689749 1.3263614
4 0.07050839 0.6603038 1.4681936 2.1990057
5 0.12928774 -0.2140063 0.4425564 0.3578378
6 1.71506499 0.1928270 0.1462031 2.0540952O problema é que se tivéssemos, por exemplo, 1000 colunas, não seria prático escrever a soma incluindo cada uma delas. Além disso, em alguns casos temos funções específicas que queremos aplicar a cada uma das linhas, o que nos obriga a encontrar uma outras formas de lidar com essa questão.
Solução 1: for loop
Podemos criar um for loop para inserir a soma de cada linha na coluna D. Primeiro, temos que lembrar que o objetivo deste post é avaliar as diferenças de desempenho entre o for loop e seus possíveis substitutos. Então, além de mostrar abaixo como fazer o for loop, vamos também calcular o tempo que a tarefa demora.
Para realizar o for loop, usamos uma variável i que varia de 1 até o número de linhas que o nosso data frame possui. Tal variável é responsável por selecionar a linha específica que queremos somar e também por indicar o local no qual a soma será inserida no data frame.
# Criar coluna com NA
dados_base_loop <- dados_base %>% mutate(D = NA)
# Realizar o loop e medir o tempo gasto
loop_time <- system.time({
for (i in 1:nrow(dados_base)) {
dados_base_loop$D[i] <- sum(dados_base_loop[i, 1:3])
}
})
head(dados_base_loop)
A B C D
1 -0.56047565 0.2620001 -1.0080707 -1.3065463
2 -0.23017749 -1.5209594 1.3549394 -0.3961975
3 1.55870831 0.2366280 -0.4689749 1.3263614
4 0.07050839 0.6603038 1.4681936 2.1990057
5 0.12928774 -0.2140063 0.4425564 0.3578378
6 1.71506499 0.1928270 0.1462031 2.0540952
loop_time
user system elapsed
492.14 279.67 774.86 Podemos ver que o data frame criado é o que realmente queríamos gerar (basta comparar a coluna D deste data frame e do data frame da seção anterior) e que o tempo necessário para a realização dessa tarefa foi 774.86 segundos.
Solução 2: foreach
Basicamente, esta abordagem é a mesma da anterior, mas neste caso paralelizando o processo. Isto quer dizer que mais de um núcleo do computador realiza a mesma tarefa. Entretanto, cada um dos núcleos atua em partes diferentes do data frame.
library(foreach)
library(doParallel)
# configurar número de núcleos que serão utilizados
cores=detectCores()
cl <- makeCluster(3) # usar 3 núcleos
registerDoParallel(cl)
# Realizar o loop e medir o tempo gasto
foreach_time <- system.time({
D_valores_foreach <- foreach (i = 1:nrow(dados_base)) %dopar% {
return(sum(dados_base[i,]))
}
dados_base_foreach <- dados_base
dados_base_foreach$D <- D_valores_foreach
})
stopCluster(cl)
head(dados_base_foreach)
A B C D
1 -0.56047565 0.2620001 -1.0080707 -1.306546
2 -0.23017749 -1.5209594 1.3549394 -0.3961975
3 1.55870831 0.2366280 -0.4689749 1.326361
4 0.07050839 0.6603038 1.4681936 2.199006
5 0.12928774 -0.2140063 0.4425564 0.3578378
6 1.71506499 0.1928270 0.1462031 2.054095
foreach_time
user system elapsed
192.28 16.84 216.53 A coluna D gerada é como esperávamos. Além disso, o tempo total para realização da tarefa reduz consideravelmente para 216.53 segundos.
Solução 3: família apply
Primeiramente, vale uma breve explicação do que é a família apply. A família apply é composta de funções que pertence ao R base as quais aplicam outras funções a determinadas estrutura. Claro? Provavelmente não. Vamos resumir algumas dessas funções para ficar mais fácil de entender:
apply()age em arrays (matrizes ou objetos similares a matriz com mais de duas dimensões). É útil quando queremos aplicar a mesma função às linhas ou colunas de uma matriz;lapply()age em listas, data frames e vetores. Aplica uma função a todos elementos de uma lista, vetor, etc. Retorna uma lista contendo os resultados.sapply()é similar aolapply(), mas tenta retornar um objeto mais simples do que uma lista, como um vetor por exemplo.mapply()permite utilizar funções que aceitam apenas escalares, mas funciona de maneira similar aosapply().
Ok, mas como utilizar a família apply na nossa tarefa? Pelas definições acima, a melhor função para o nosso caso é apply(). No nosso caso, esta função retornará um vetor com valores da coluna D. Então uma tarefa a mais será necessária: concatenar este vetor ao data frame final. Vamos ao exemplo:
apply_time <- system.time({
D_valores_apply <- apply(X = dados_base, MARGIN = 1, FUN = sum)
dados_base_apply <- dados_base
dados_base_apply$D <- D_valores_apply
})
head(dados_base_apply)
A B C D
1 -0.56047565 0.2620001 -1.0080707 -1.3065463
2 -0.23017749 -1.5209594 1.3549394 -0.3961975
3 1.55870831 0.2366280 -0.4689749 1.3263614
4 0.07050839 0.6603038 1.4681936 2.1990057
5 0.12928774 -0.2140063 0.4425564 0.3578378
6 1.71506499 0.1928270 0.1462031 2.0540952
apply_time
user system elapsed
0.76 0.00 0.77 O argumento X é a matriz na qual aplicamos a função FUN, no caso sum. O argumento MARGIN diz em qual direção queremos aplicar a nossa função. 1 quer dizer que será aplicado a função sum em cada linha da matrix X e 2 em cada coluna da mesma matriz.
Usando a função apply(), o novo tempo para realizar a tarefa é de 0.77 segundos. Ótimo aumento de desempenho, não é mesmo?
Solução 4: future apply
Usando o pacote future.apply é possível paralelizar o processo anterior que usou apply.
library(future.apply)
plan(multiprocess, workers = 3) ## Paralelização usando 3 núcleos
futureapply_time <- system.time({
D_valores_futureapply <- future_apply(X = dados_base, MARGIN = 1, FUN = sum)
dados_base_futureapply <- dados_base
dados_base_futureapply$D <- D_valores_futureapply
})
head(dados_base_futureapply)
A B C D
1 -0.56047565 0.2620001 -1.0080707 -1.3065463
2 -0.23017749 -1.5209594 1.3549394 -0.3961975
3 1.55870831 0.2366280 -0.4689749 1.3263614
4 0.07050839 0.6603038 1.4681936 2.1990057
5 0.12928774 -0.2140063 0.4425564 0.3578378
6 1.71506499 0.1928270 0.1462031 2.0540952
futureapply_time
user system elapsed
5.53 0.08 6.85 Notamos que neste caso não houve melhoria de desempenho. Nem sempre a paralelização melhora a performance. Isso ocorre porque ao paralelizar a tarefa, é necessário que haja sincronização dos cores em alguns momentos para inserir os resultados na posição correta e isso pode gerar um aumento no tempo gasto.
Solução 5: purrr
Vamos testar uma outra alternativa, o pacote purrr. Esse pacote é parte de um pacote mais abrangente chamado tidyverse. E essa é uma vantagem das funções do purrr por conseguir interagir facilmente com os demais pacote do tidyverse.
Para a realização da tarefas que devemos realizar, vamos utilizar a função pmap(), que é uma alternativa para realização de iterações em linhas de um data frame. No final das iterações, uma lista com um elemento para cada linha será retornado. Por isso, alguma manipulações extras nos dados são necessárias.
Vamos aos testes:
library(purrr)
purrr_time <- system.time({
D_valores_map <- pmap(dados_base, sum) %>% unlist()
dados_base_map <- dados_base
dados_base_map$D <- D_valores_map
})
head(dados_base_map)
A B C D
1 -0.56047565 0.2620001 -1.0080707 -1.3065463
2 -0.23017749 -1.5209594 1.3549394 -0.3961975
3 1.55870831 0.2366280 -0.4689749 1.3263614
4 0.07050839 0.6603038 1.4681936 2.1990057
5 0.12928774 -0.2140063 0.4425564 0.3578378
6 1.71506499 0.1928270 0.1462031 2.0540952
purrr_time
user system elapsed
1.94 0.00 1.94 usando o purrr, obtivemos um resultado levemente pior (1.94 segundos) do que no caso do apply. Porém, quando comparado com for loop, foreach e future.apply, o purrr realizou a tarefa mais rapidamente.
Solução 6: furrr
Finalmente, vamos paralelizar a solução anterior usando o pacote furrr. Ele funciona de forma muito similar ao purrr, apenas trocando a função pmap() por future_pmap(), como pode ser visto abaixo.
library(furrr)
plan(multiprocess)
furrr_time <- system.time({
D_valores_furr <- future_pmap(dados_base, sum) %>% unlist()
dados_base_furr <- dados_base
dados_base_furr$D <- D_valores_furr
})
head(dados_base_furr)
A B C D
1 -0.56047565 0.2620001 -1.0080707 -1.3065463
2 -0.23017749 -1.5209594 1.3549394 -0.3961975
3 1.55870831 0.2366280 -0.4689749 1.3263614
4 0.07050839 0.6603038 1.4681936 2.1990057
5 0.12928774 -0.2140063 0.4425564 0.3578378
6 1.71506499 0.1928270 0.1462031 2.0540952
furrr_time
user system elapsed
0.20 0.00 1.98 Houve uma leve perda de desempenho em relação ao purrr and apply, mas o resultado foi melhor do que a demais soluções.
Conclusão
Em uma simples tarefa de criação de uma nova coluna em um data frame baseada em informações das demais colunas, testamos uma abordagem usando for loop e algumas alternativas a ele. Vimos que, neste caso (para outro tipo de tarefas os resultados pode ser bastante diferentes), o for loop foi a alternativa de pior desempenho (ver gráfico abaixo).
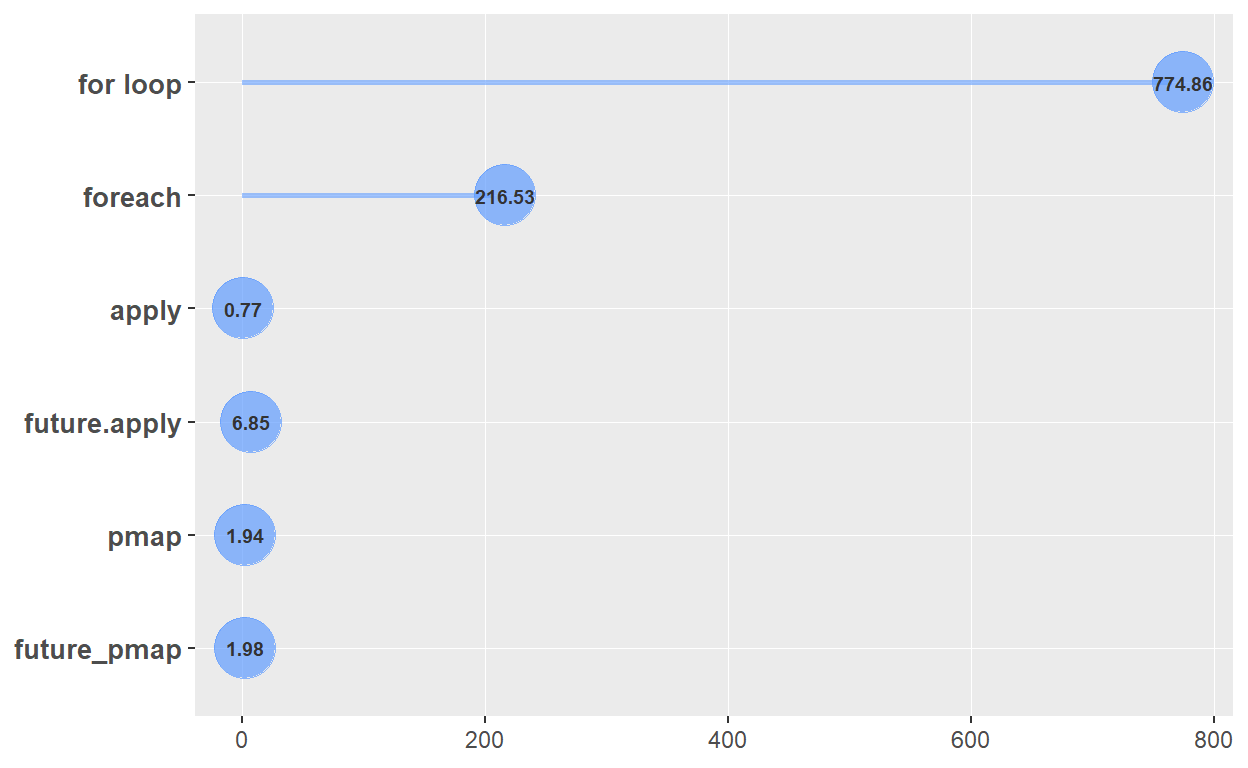
Além disso, com exceção do for loop, as estratégias de paralelização não aumentaram a velocidade de execução da tarefa, apesar de haver diversos exemplos mostrando casos em que tais estratégias podem ser utilizadas de forma bastante efetiva. Neste caso, o custo de distribuição da tarefa entre os núcleos (workers) é maior do que o ganho de tempo por dividir a tarefa entre eles. Vamos apresentar em outro post o processo de paralelização de forma mais detalhada e incluir exemplos nos quais a paralelização apresenta ganhos significativos de desempenho. De todo modo, o objetivo deste post foi alcançado: mostramos que existem alternativas ao for loop que trazem um relevante aumento de performance.